10 - 匹配法#
回归到底在做什么?#
正如我们目前所看到的,当我们进行处理组与对照组比较时,回归在控制额外变量方面表现得非常出色。若我们具备独立性 \((Y_0, Y_1)\perp T | X\),那么回归能通过控制 X 来识别平均处理效应(ATE)。回归实现这一过程的方式近乎神奇。为获得直观理解,让我们回顾所有 X 变量均为虚拟变量的情形。此时,回归将数据划分至各虚拟单元,并计算处理组与对照组间的均值差异。由于是在固定的 X 虚拟单元内进行,这种均值差异能保持 X 恒定。这相当于执行 \(E[Y|T=1] - E[Y|T=0] | X=x\),其中 \(x\) 代表一个虚拟单元(例如所有虚拟变量设为 1)。随后,回归通过加权整合各单元估计值以生成最终 ATE,其权重分配与各处理组内处理变量的方差成比例。
Show code cell source
import warnings
warnings.filterwarnings('ignore')
import pandas as pd
import numpy as np
from matplotlib import style
from matplotlib import pyplot as plt
import statsmodels.formula.api as smf
import graphviz as gr
%matplotlib inline
style.use("fivethirtyeight")
举个例子,假设我正在估计一种药物的效果,并且我有6名男性和4名女性。我的因变量是住院天数,我希望这款药物能缩短住院时间。对于男性,药物的真实因果效应是 -3,也就是说药物可以使他们的住院时间减少3天;而对女性,效应是 -2。
让情况更复杂一点,男性受这种疾病影响更严重,因此住院时间更长,并且他们更有可能接受这种药物——6个男性中只有1人没有服药。相对地,女性对这种疾病的抵抗力更强,住院时间也更短,且她们中有50%的人服用了药物。
drug_example = pd.DataFrame(dict(
sex= ["M","M","M","M","M","M", "W","W","W","W"],
drug=[1,1,1,1,1,0, 1,0,1,0],
days=[5,5,5,5,5,8, 2,4,2,4]
))
注意,简单地比较处理组和对照组会得出一个带有负偏误的估计结果,也就是说,药物看起来没有它实际那么有效。这是可以预期的,因为我们遗漏了“性别”这个混杂变量。在这种情况下,估计的平均处理效应(ATE)小于真实值,因为男性更可能接受药物治疗,同时也更容易受到疾病的影响。
drug_example.query("drug==1")["days"].mean() - drug_example.query("drug==0")["days"].mean()
-1.1904761904761898
鉴于男性真实效应为-3 而女性为-2,平均处理效应(ATE)应为
\( ATE=\dfrac{(-3*6) + (-2*4)}{10}=-2.6 \)
这个估计是通过以下三个步骤完成的:1)将数据按混杂变量划分为不同的单元,在这个例子中就是男性组和女性组;2)在每个单元内分别估计处理效应;3)将这些效应按加权平均进行组合,其中权重是每个单元或协变量组的样本量。如果我们的数据中男性和女性的数量完全相等,那么估计出的平均处理效应(ATE)就会正好是两组效应的中间值,也就是 -2.5。由于数据中男性多于女性,最终的ATE估计会更接近男性组的效应值。这种估计方法被称为非参数估计,因为它对数据的生成过程没有做出任何特定假设。
如果我们使用回归来控制性别变量,就等于在模型中加入了线性关系的假设。回归同样会将数据划分为男性和女性两个组,并分别估计它们各自的效应。这一切看起来都没问题。然而,在将各组效应合并时,回归并不是根据样本数量加权的。相反,回归使用的权重与各组中处理变量的方差成正比。在我们的例子中,由于只有一个男性属于对照组,男性组中处理变量的方差小于女性组。更具体地说,男性的处理变量 T 的方差为 \(0.139=1/6*(1 - 1/6)\) ,而女性的为 \(0.25=2/4*(1 - 2/4)\)。因此,在这个例子中,回归会对女性组给予更高的权重,最终估计的 ATE 会更接近女性的效应值 -2。
smf.ols('days ~ drug + C(sex)', data=drug_example).fit().summary().tables[1]
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 7.5455 | 0.188 | 40.093 | 0.000 | 7.100 | 7.990 |
| C(sex)[T.W] | -3.3182 | 0.176 | -18.849 | 0.000 | -3.734 | -2.902 |
| drug | -2.4545 | 0.188 | -13.042 | 0.000 | -2.900 | -2.010 |
虚拟变量能更直观呈现该结果,但回归分析以其独特方式在估算效应时同样保持连续变量的恒定。对于连续变量,平均处理效应(ATE)也会向协变量方差更大的方向偏移。
由此我们了解到回归分析有其独特性。它是线性的、参数化的,偏好高方差特征……这既可能是优点也可能是缺点,取决于具体情境。正因如此,掌握其他控制混杂因素的技术至关重要。这些方法不仅是因果分析工具箱中的额外工具,理解处理混杂因素的不同方式更能拓宽我们对问题的认知。基于这一考量,现在我要向各位介绍子类估计量!
子类估计量#
当我们需要估计某项因果效应(如职业培训对收入的影响)而处理变量并非随机分配时,必须警惕混杂因素。可能存在只有积极性更高的人群才会参加培训的情况,且无论是否参加培训这些人群原本就具有更高的收入水平。我们需要在动机水平及其他潜在混杂因素基本相似的小群体中,评估培训项目的真实效果。
更一般地说,如果我们想估计某种因果效应,但由于某些变量 X 的混杂而难以实现,我们需要做的是在 X 相同的小群体内进行处理组与对照组的比较。若我们具备条件独立性 \((Y_0, Y_1)\perp T | X\),则可按下式表达平均处理效应(ATE)。
\( ATE = \int(E[Y|X, T=1] - E[Y|X, T=0])dP(x) \)
该积分的作用是遍历特征 X 分布的所有空间,计算所有这些微小空间内的均值差异,并将所有结果综合为平均处理效应。另一种理解方式是考虑一组离散特征。此时,我们可以说特征 X 分布在 K 个不同的单元 \(\{X_1, X_2, ..., X_k\}\) 中,我们所做的是计算每个单元内的处理效应,并将它们合并为 ATE。在此离散情形下,将积分转换为求和,即可推导出子类估计量。
\( \hat{ATE} = \sum^K_{k=1}(\bar{Y}_{k1} - \bar{Y}_{k0}) * \dfrac{N_k}{N} \)
其中横线代表处理组结果的平均值 \(Y_{k1}\), 与非处理组 \(Y_{k0}\) 在第 k 个单元中的均值,\(N_{k}\) 表示该单元内的观测数量。可以看出,我们为每个单元计算局部平均处理效应(ATE),并通过加权平均进行合并,权重即为各单元的样本量。在上述医学案例中,该方法的首次估计值为−2.6。
匹配估计量#

子类估计量在实际中应用较少(稍后将揭示原因——维度灾难问题),但它清晰展示了因果推断估计量应具备的功能:如何有效控制混杂变量。这为我们探索其他类型的估计量(如匹配估计量)提供了理论基础。
其核心理念高度相似。由于某些混杂变量 X 导致处理组与对照组初始不可比,可通过为每个处理单元匹配相似的对照单元实现可比性。这相当于为每个处理单元寻找未处理的”双胞胎”。通过这种配对比较,处理组与对照组重新获得可比性。
举例而言,假设我们正试图评估一项培训项目对收入的影响。以下是受训者的基本情况
trainee = pd.read_csv("./data/trainees.csv")
trainee.query("trainees==1")
| unit | trainees | age | earnings | |
|---|---|---|---|---|
| 0 | 1 | 1 | 28 | 17700 |
| 1 | 2 | 1 | 34 | 10200 |
| 2 | 3 | 1 | 29 | 14400 |
| 3 | 4 | 1 | 25 | 20800 |
| 4 | 5 | 1 | 29 | 6100 |
| 5 | 6 | 1 | 23 | 28600 |
| 6 | 7 | 1 | 33 | 21900 |
| 7 | 8 | 1 | 27 | 28800 |
| 8 | 9 | 1 | 31 | 20300 |
| 9 | 10 | 1 | 26 | 28100 |
| 10 | 11 | 1 | 25 | 9400 |
| 11 | 12 | 1 | 27 | 14300 |
| 12 | 13 | 1 | 29 | 12500 |
| 13 | 14 | 1 | 24 | 19700 |
| 14 | 15 | 1 | 25 | 10100 |
| 15 | 16 | 1 | 43 | 10700 |
| 16 | 17 | 1 | 28 | 11500 |
| 17 | 18 | 1 | 27 | 10700 |
| 18 | 19 | 1 | 28 | 16300 |
以下是非培训人员:
trainee.query("trainees==0")
| unit | trainees | age | earnings | |
|---|---|---|---|---|
| 19 | 20 | 0 | 43 | 20900 |
| 20 | 21 | 0 | 50 | 31000 |
| 21 | 22 | 0 | 30 | 21000 |
| 22 | 23 | 0 | 27 | 9300 |
| 23 | 24 | 0 | 54 | 41100 |
| 24 | 25 | 0 | 48 | 29800 |
| 25 | 26 | 0 | 39 | 42000 |
| 26 | 27 | 0 | 28 | 8800 |
| 27 | 28 | 0 | 24 | 25500 |
| 28 | 29 | 0 | 33 | 15500 |
| 29 | 31 | 0 | 26 | 400 |
| 30 | 32 | 0 | 31 | 26600 |
| 31 | 33 | 0 | 26 | 16500 |
| 32 | 34 | 0 | 34 | 24200 |
| 33 | 35 | 0 | 25 | 23300 |
| 34 | 36 | 0 | 24 | 9700 |
| 35 | 37 | 0 | 29 | 6200 |
| 36 | 38 | 0 | 35 | 30200 |
| 37 | 39 | 0 | 32 | 17800 |
| 38 | 40 | 0 | 23 | 9500 |
| 39 | 41 | 0 | 32 | 25900 |
若进行简单的均值比较,我们会发现受训者的收入低于未参与该项目的人员。
trainee.query("trainees==1")["earnings"].mean() - trainee.query("trainees==0")["earnings"].mean()
-4297.49373433584
然而,观察上表可见,受训者年龄普遍小于非受训者,这表明年龄可能是一个混淆变量。为此,我们将采用年龄匹配法进行调整。具体操作如下:将处理组的第 1 单元与非处理组的第 27 单元配对(两者均为 28 岁),第 2 单元与第 34 单元配对,第 3 单元与第 37 单元配对,第 4 单元则与第 35 单元配对……当处理到第 5 单元时,需从非处理组中寻找同为 29 岁的匹配对象,但符合条件的第 37 单元已被使用。这并不构成问题,因为同一单元可重复使用。若存在多个匹配单元,则可随机选择其中之一进行配对。
这是前 7 个单元的匹配数据集样貌
# make dataset where no one has the same age
unique_on_age = (trainee
.query("trainees==0")
.drop_duplicates("age"))
matches = (trainee
.query("trainees==1")
.merge(unique_on_age, on="age", how="left", suffixes=("_t_1", "_t_0"))
.assign(t1_minuts_t0 = lambda d: d["earnings_t_1"] - d["earnings_t_0"]))
matches.head(7)
| unit_t_1 | trainees_t_1 | age | earnings_t_1 | unit_t_0 | trainees_t_0 | earnings_t_0 | t1_minuts_t0 | |
|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 1 | 28 | 17700 | 27 | 0 | 8800 | 8900 |
| 1 | 2 | 1 | 34 | 10200 | 34 | 0 | 24200 | -14000 |
| 2 | 3 | 1 | 29 | 14400 | 37 | 0 | 6200 | 8200 |
| 3 | 4 | 1 | 25 | 20800 | 35 | 0 | 23300 | -2500 |
| 4 | 5 | 1 | 29 | 6100 | 37 | 0 | 6200 | -100 |
| 5 | 6 | 1 | 23 | 28600 | 40 | 0 | 9500 | 19100 |
| 6 | 7 | 1 | 33 | 21900 | 29 | 0 | 15500 | 6400 |
注意最后一列显示的是处理组与其匹配的未处理组在收入上的差异。若取该列的平均值,我们便得到在控制年龄因素后的平均处理效应估计值(ATET)。与之前仅采用简单均值差的方法相比,可见此时的估计结果显著偏向正值。
matches["t1_minuts_t0"].mean()
2457.8947368421054
但这只是个刻意设计的示例,仅为引入匹配概念。现实中,我们通常面对多个特征且单元间无法完美匹配的情形。此时,需定义某种邻近度度量来比较单元间的相似程度。常用的度量之一是欧几里德范数 \(||X_i - X_j||\)。然而,该差异对特征尺度并不具有不变性。这意味着像年龄这样取值在十这个量级的特征,在计算范数时其重要性将远低于收入这类百位量级的特征。因此,在应用范数前,需对特征进行缩放处理,使它们大致处于同一量级。
在定义了距离度量之后,我们现在可以将匹配定义为与待匹配样本最接近的邻居。用数学术语表达,匹配估计量可以写作如下形式
\( \hat{ATE} = \frac{1}{N} \sum^N_{i=1} (2T_i - 1)\big(Y_i - Y_{jm}(i)\big) \)
其中 \(Y_{jm}(i)\) 代表来自另一处理组中与 \(Y_i\) 最为相似的样本。我们进行 \(2T_i - 1\) 双向匹配:既将处理组与对照组匹配,也将对照组与处理组匹配。
为验证该估计量,我们以药物为例进行说明。再次强调,我们的目标是探究药物对康复天数的影响。然而,该效应受到病情严重程度、性别及年龄的混杂影响。我们有理由相信,病情更严重的患者接受药物治疗的概率更高。
med = pd.read_csv("./data/medicine_impact_recovery.csv")
med.head()
| sex | age | severity | medication | recovery | |
|---|---|---|---|---|---|
| 0 | 0 | 35.049134 | 0.887658 | 1 | 31 |
| 1 | 1 | 41.580323 | 0.899784 | 1 | 49 |
| 2 | 1 | 28.127491 | 0.486349 | 0 | 38 |
| 3 | 1 | 36.375033 | 0.323091 | 0 | 35 |
| 4 | 0 | 25.091717 | 0.209006 | 0 | 15 |
如果我们观察一个简单的均值差异 \(E[Y|T=1]-E[Y|T=0]\),会发现接受治疗的患者平均比未治疗者多需要 16.9 天恢复。这很可能是由于混杂因素造成的,因为我们并不预期药物会对患者造成伤害。
med.query("medication==1")["recovery"].mean() - med.query("medication==0")["recovery"].mean()
16.895799546498726
为了纠正这种偏误,我们将通过匹配来控制变量 X。首先,需要记得对特征进行标准化处理,否则在计算点间距离时,像年龄这样的特征会比严重程度等特征具有更高权重。为此,我们可以对特征进行标准化。
# scale features
X = ["severity", "age", "sex"]
y = "recovery"
med = med.assign(**{f: (med[f] - med[f].mean())/med[f].std() for f in X})
med.head()
| sex | age | severity | medication | recovery | |
|---|---|---|---|---|---|
| 0 | -0.996980 | 0.280787 | 1.459800 | 1 | 31 |
| 1 | 1.002979 | 0.865375 | 1.502164 | 1 | 49 |
| 2 | 1.002979 | -0.338749 | 0.057796 | 0 | 38 |
| 3 | 1.002979 | 0.399465 | -0.512557 | 0 | 35 |
| 4 | -0.996980 | -0.610473 | -0.911125 | 0 | 15 |
现在,进入匹配本身。我们不会编写匹配函数,而是使用 Sklearn 中的 K 最近邻算法。该算法通过寻找估计或训练集中最近的数据点来进行预测。
在匹配过程中,我们需要其中的两个。其一, mt0 ,将存储未经处理的点,并在被要求时在未处理数据中寻找匹配项。另一个, mt1 ,则存储经过处理的点,并在需要时在已处理数据中寻找匹配项。完成此拟合步骤后,我们可以利用这些 KNN 模型进行预测,这些预测即为我们的匹配结果。
from sklearn.neighbors import KNeighborsRegressor
treated = med.query("medication==1")
untreated = med.query("medication==0")
mt0 = KNeighborsRegressor(n_neighbors=1).fit(untreated[X], untreated[y])
mt1 = KNeighborsRegressor(n_neighbors=1).fit(treated[X], treated[y])
predicted = pd.concat([
# find matches for the treated looking at the untreated knn model
treated.assign(match=mt0.predict(treated[X])),
# find matches for the untreated looking at the treated knn model
untreated.assign(match=mt1.predict(untreated[X]))
])
predicted.head()
| sex | age | severity | medication | recovery | match | |
|---|---|---|---|---|---|---|
| 0 | -0.996980 | 0.280787 | 1.459800 | 1 | 31 | 39.0 |
| 1 | 1.002979 | 0.865375 | 1.502164 | 1 | 49 | 52.0 |
| 7 | -0.996980 | 1.495134 | 1.268540 | 1 | 38 | 46.0 |
| 10 | 1.002979 | -0.106534 | 0.545911 | 1 | 34 | 45.0 |
| 16 | -0.996980 | 0.043034 | 1.428732 | 1 | 30 | 39.0 |
有了这些匹配项,我们现在可以应用匹配估计器公式
\( \hat{ATE} = \frac{1}{N} \sum^N_{i=1} (2T_i - 1)\big(Y_i - Y_{jm}(i)\big) \)
np.mean((2*predicted["medication"] - 1)*(predicted["recovery"] - predicted["match"]))
-0.9954
通过这种匹配方式,我们可以看出药物的效果已不再积极。这意味着,在控制变量 X 的情况下,该药物平均能将康复时间缩短约 1 天。相较于原先存在偏误、预测康复时间会增加 16.9 天的估计,这已然是一个巨大的进步。
然而,我们还能做得更好。
匹配偏误#
我们发现,如上设计的匹配估计量实际上是有偏的。为了理解这一点,让我们考虑 ATET(处理组平均处理效应)估计量而非 ATE(平均处理效应),仅因其表达更为简洁。这一直觉同样适用于 ATE。
\( \hat{ATET} = \frac{1}{N_1}\sum (Y_i - Y_j(i)) \)
其中 \(N_1\) 代表接受处理的个体数量,\(Y_j(i)\) 为处理单元 i 的未处理匹配项。为检验偏误,我们所做的是期望能够应用中心极限定理,使得下方内容收敛于均值为零的正态分布。
\( \sqrt{N_1}(\hat{ATET} - ATET) \)
然而,这并非总是发生。若我们定义给定 X 下未处理组的平均结果为 \(\mu_0(x)=E[Y|X=x, T=0]\),则有(顺便提一下,此处我省略了证明过程,因其稍偏离主题)。
\( E[\sqrt{N_1}(\hat{ATET} - ATET)] = E[\sqrt{N_1}(\mu_0(X_i) - \mu_0(X_j(i)))] \)
现在,\(\mu_0(X_i) - \mu_0(X_j(i))\) 并不那么容易理解,因此我们需要更仔细地审视它。\(\mu_0(X_i)\) 是处理单元 \(i\) 若未接受处理时的结果 Y 值,即单元 i 的反事实结果 \(Y_0\)。\(\mu_0(X_j(i))\) 则是未处理单元 \(j\) ——即单元 \(i\) 的匹配对象——的结果。因此,它同样是 \(Y_0\) ,但这次针对的是单元 \(j\)。只不过这一次是实际结果,因为 \(j\) 属于未处理组。由于 \(j\) 与 \(i\) 仅是相似而非完全相同,这一差值很可能不为零。换言之,\(X_i \approx X_j \)。所以,\(Y_{0i} \approx Y_{0j} \)。
随着样本量的增加,可匹配的单元数量增多,单元 \(i\) 与其匹配单元 \(j\) 之间的差异也会缩小。但这种差异收敛至零的速度较慢。因此,\(E[\sqrt{N_1}(\mu_0(X_i) - \mu_0(X_j(i)))]\) 可能不会收敛至零,因为 \(\sqrt{N_1}\) 的增长速度快于 \((\mu_0(X_i) - \mu_0(X_j(i)))\) 的减小速度。
当匹配差异巨大时,偏误便会产生。幸运的是,我们知晓如何修正这一问题。每项观测数据对偏误的贡献为 \((\mu_0(X_i) - \mu_0(X_j(i)))\) ,因此只需在估计量中从每次匹配比较中减去这一数值即可。为此,我们可以用该数值 \(\mu_0(X_j(i))\) 的某种估计量(如通过线性回归等模型获得)来替代 \(\hat{\mu_0}(X_j(i))\)。这将 ATET 估计量更新为以下方程式
\( \hat{ATET} = \frac{1}{N_1}\sum \big((Y_i - Y_{j(i)}) - (\hat{\mu_0}(X_i) - \hat{\mu_0}(X_{j(i)}))\big) \)
其中 \(\hat{\mu_0}(x)\) 为 \(E[Y|X, T=0]\) 的某种估计值,例如仅基于未处理样本拟合的线性回归结果
from sklearn.linear_model import LinearRegression
# fit the linear regression model to estimate mu_0(x)
ols0 = LinearRegression().fit(untreated[X], untreated[y])
ols1 = LinearRegression().fit(treated[X], treated[y])
# find the units that match to the treated
treated_match_index = mt0.kneighbors(treated[X], n_neighbors=1)[1].ravel()
# find the units that match to the untreatd
untreated_match_index = mt1.kneighbors(untreated[X], n_neighbors=1)[1].ravel()
predicted = pd.concat([
(treated
# find the Y match on the other group
.assign(match=mt0.predict(treated[X]))
# build the bias correction term
.assign(bias_correct=ols0.predict(treated[X]) - ols0.predict(untreated.iloc[treated_match_index][X]))),
(untreated
.assign(match=mt1.predict(untreated[X]))
.assign(bias_correct=ols1.predict(untreated[X]) - ols1.predict(treated.iloc[untreated_match_index][X])))
])
predicted.head()
| sex | age | severity | medication | recovery | match | bias_correct | |
|---|---|---|---|---|---|---|---|
| 0 | -0.996980 | 0.280787 | 1.459800 | 1 | 31 | 39.0 | 4.404034 |
| 1 | 1.002979 | 0.865375 | 1.502164 | 1 | 49 | 52.0 | 12.915348 |
| 7 | -0.996980 | 1.495134 | 1.268540 | 1 | 38 | 46.0 | 1.871428 |
| 10 | 1.002979 | -0.106534 | 0.545911 | 1 | 34 | 45.0 | -0.496970 |
| 16 | -0.996980 | 0.043034 | 1.428732 | 1 | 30 | 39.0 | 2.610159 |
随即产生的一个直接问题是:这样做难道不会违背匹配的初衷吗?如果无论如何我都得运行线性回归,为何不直接使用它,而要采用这个复杂的模型。这是个合理的质疑,因此我需要花些时间来解答。
首先,我们所拟合的线性回归并不通过外推处理维度来获取处理效应。相反,其目的仅在于校正偏误。此处的线性回归具有局部性,即它并不试图考察若处理组看起来像未处理组时会如何。它完全不进行此类外推。这部分工作留给了匹配环节。估计量的核心仍是匹配部分。我想在此强调的是,对于这一估计量而言,普通最小二乘法(OLS)处于次要地位。
第二点在于,匹配是一种非参数估计方法。它不假设线性或任何参数模型。因此,相较于线性回归,匹配更具灵活性,能在非线性特征极其显著而线性回归失效的场景中发挥作用。
这是否意味着你只应使用匹配法?嗯,这是个棘手的问题。阿尔贝托·阿巴迪主张确实应该如此,认为这种方法更为灵活,且一旦掌握了代码,操作起来同样简单。我对此并不完全信服。举例来说,阿巴迪投入了大量时间研究并开发这一估计方法(没错,他是推动匹配法发展至今日形态的科学家之一),显然他对这种方法有着个人情感上的投入。其次,线性回归的简洁性中有某些特质是匹配法所不具备的。“保持其他条件不变”的偏导数数学原理,在线性回归中比在匹配法中更易于理解。但这仅是我的个人偏好。老实说,这个问题并没有明确的答案。无论如何,回到我们的例子中来。
使用偏误校正公式后,我得到了如下的平均处理效应(ATE)估计值。
np.mean((2*predicted["medication"] - 1)*((predicted["recovery"] - predicted["match"])-predicted["bias_correct"]))
-7.362660906141391
当然,我们还需要围绕这一测量值设置置信区间,但数学理论就暂且讨论到这里。实际操作中,我们可以直接借用他人编写的代码,导入一个匹配估计器。比如,这是来自因果推断库 causalinference 中的一个例子。
from causalinference import CausalModel
cm = CausalModel(
Y=med["recovery"].values,
D=med["medication"].values,
X=med[["severity", "age", "sex"]].values
)
cm.est_via_matching(matches=1, bias_adj=True)
print(cm.estimates)
Treatment Effect Estimates: Matching
Est. S.e. z P>|z| [95% Conf. int.]
--------------------------------------------------------------------------------
ATE -7.709 0.609 -12.649 0.000 -8.903 -6.514
ATC -6.665 0.246 -27.047 0.000 -7.148 -6.182
ATT -9.679 1.693 -5.717 0.000 -12.997 -6.361
最终,我们可以有把握地说,我们的药物确实缩短了患者住院时间。由于 knn sklearn 实现与 causalinference Python 包在匹配平局处理上的差异,ATE 估计值略低于我的计算结果。
在结束这个话题之前,我想再稍微讨论一下匹配偏误的成因。我们发现当个体与其匹配对象相似度不足时,匹配会产生偏误。但究竟是什么导致它们如此不同?
纬度灾难#
事实证明,答案既简单又直观。基于少数特征(如性别)匹配个体很容易,但当我们增加年龄、收入、出生城市等更多特征时,寻找匹配对象就变得越来越困难。更概括地说,特征维度越高,个体与其匹配对象之间的距离就越大。
这不仅影响了匹配估计器的表现,还与之前提到的子类估计器密切相关。回顾早期那个关于药物效果在男女性别上差异的人为设计例子,构建子分类估计器相对简单,因为我们仅有两个类别:男性和女性。然而,若类别增多呢?假设我们有两个连续变量,如年龄和收入,并将它们各自离散化为 5 个区间,这将生成 25 个单元格,即 \(5^2\)。更进一步,如果有 10 个协变量,每个分 3 个区间,看似不多,对吧?但实际上,这将产生 59049 个单元格,即 \(3^{10}\)。显而易见,问题规模会迅速膨胀。这一现象在数据科学中普遍存在,被称为 “维度灾难” !!!
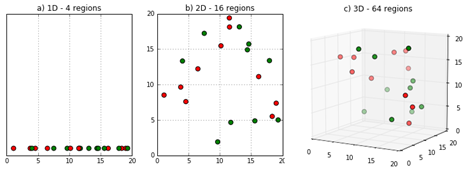 图片来源: https://deepai.org/machine-learning-glossary-and-terms/curse-of-dimensionality
图片来源: https://deepai.org/machine-learning-glossary-and-terms/curse-of-dimensionality
尽管这个名称听起来吓人且故作高深,但它实际上仅意味着填满特征空间所需的数据点数量会随着特征或维度的增加呈指数级增长。例如,若填满 3 个特征空间需要 X 个数据点,那么填满 4 个特征的空间则需要指数级更多的点。
在子类估计器的语境下,维度灾难意味着当特征数量庞大时,其性能将受到影响。大量特征意味着 X 中存在众多单元格。若单元格过多,部分单元格将仅有极少数据,甚至可能仅包含处理组或对照组,导致无法在该处估计平均处理效应(ATE),从而破坏估计器的有效性。在匹配情境中,这意味着特征空间将极为稀疏,单元间距离会非常遥远,这将增大匹配对之间的距离并引发偏误问题。
至于线性回归,它实际上能很好地处理这个问题。其做法是将所有特征 X 投影至单一的 Y 维度上,然后在该投影上进行处理组与对照组的比较。因此,从某种意义上说,线性回归通过某种降维方式来估计平均处理效应(ATE),这一方法相当精妙。
大多数因果模型也具备应对维度灾难的方法。虽然我不再赘述,但你在研究这些模型时应时刻牢记这一点。例如,在下一节讨论倾向得分时,不妨观察它是如何解决这一问题的。
核心要点#
本节伊始,我们理解了线性回归的功能及其如何助力识别因果关系。具体而言,我们认识到回归可视为将数据集划分为多个单元,计算每个单元内的 ATE,再将各单元的 ATE 整合为整个数据集的单一 ATE 值。
由此,我们推导出了一个基于子类的非常通用的因果推断估计量。虽然该估计量在实际应用中并不十分实用,但它为我们提供了关于如何解决因果推断估计问题的一些有趣见解。这进而引导我们探讨了匹配估计量的概念。
匹配法通过为每个处理单元寻找一个与之极为相似的非处理单元作为对照(反之亦然),从而控制混杂变量的影响。我们学习了如何利用 KNN 算法实现这一方法,以及如何通过回归校正其偏误。最后,我们比较了匹配法与线性回归的区别,认识到匹配作为一种非参数估计方法,不像线性回归那样依赖于线性假设。
最后,我们深入探讨了高维数据集带来的挑战,并分析了因果推断方法在此类数据中可能面临的困境。
参考文献#
我愿将这一系列作品视为对 Joshua Angrist、Alberto Abadie 和 Christopher Walters 杰出计量经济学课程的致敬。第一部分的大部分思想源自他们在美国经济学会授课的内容。在艰难的 2020 年,正是观看他们的课程视频让我保持了理智。
我还想引用 Angrist 的精彩著作。它们向我展示了计量经济学(他们称之为“Metrics”)不仅极为实用,而且充满乐趣。
最后还要感谢 Miguel Hernán 和 Jamie Robins 的《Causal Inference》一书。它是我在面对最棘手的因果问题时的可靠伙伴。

参与贡献#
《Causal Inference for the Brave and True》 是一本关于因果推断的开源教材,致力于以经济上可负担、认知上可理解的方式,普及这门“科学的统计基础”。全书基于 Python,仅使用自由开源软件编写,原始英文版本由 Matheus Facure 编写与维护。
本书的中文版由黄文喆与许文立助理教授合作翻译,并托管在 GitHub 中文主页。希望本地化的内容能帮助更多中文读者学习和掌握因果推断方法。
如果你觉得这本书对你有帮助,并希望支持该项目,可以前往 Patreon 支持原作者。
如果你暂时不方便进行经济支持,也可以通过以下方式参与贡献:
修正错别字
提出翻译或表达建议
反馈你未能理解的部分内容
欢迎前往英文版或中文版仓库点击 issues 区 或 中文版 issues 区 提出反馈。
最后,如果你喜欢这本书的内容,也请将其分享给可能感兴趣的朋友,并为项目在 GitHub 上点亮一颗星:英文版仓库 / 中文版仓库。